本ページでは以下のページを要約するとともに、個人的な解説も記載しています。独特な解釈をしている部分があるので、誤りなどの指摘はtwitterまでお願いします。
本ページはブログの記事として分割したものを1ページにまとめたものです。
参考ページ
Supervised learning: predicting an output variable from high-dimensional observations — scikit-learn 1.1.2 documentation
https://scikit-learn.org/stable/tutorial/statistical_inference/supervised_learning.html
閲覧日:2022年10月
教師あり学習とは
教師あり学習は観測された学習用のデータXと、「ターゲット」や「ラベル」とよばれる、学習データに紐づく予測しようとしているデータyの2つのデータをセットで学習します。
ほとんどの場合、ラベルは1次元配列になります。
教師あり学習で使用するestimatorは、モデルを学習させるためのメソッドfit(X,y)と、学習データXを与えたときに予測されたラベルyを返すメソッドpredict(X)を持ちます。
scikit-learn で分類を行う場合、ラベルyは整数または文字列の配列です。
最近傍と次元の呪い
アヤメの分類:
アヤメのデータセットは、花弁とがく片の長さと幅から 3 種類のアヤメ (Setosa、Versicolour、Virginica) に分類するタスクです。
以下のコードでデータの詳細を確認できます。
アヤメの種類を0,1,2の数値で表しています。
import numpy as np
from sklearn import datasets
iris_X, iris_y = datasets.load_iris(return_X_y=True)
np.unique(iris_y)
#array([0, 1, 2])
※PCAで3次元に学習データを変換させたのち、データを可視化すると以下のようになります。オリジナルコードなので汚くてすみません。
from sklearn.decomposition import PCA
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
#主成分分析の実行
pca = PCA()
pca.fit(iris_X)
score = pd.DataFrame(pca.transform(iris_X))
x = score[0]
y = score[1]
z = score[2]
fig = plt.figure()
ax = fig.add_subplot(projection='3d')
for i in range(iris_y.size):
if iris_y[i] == 0:
ax.scatter(x[i], y[i], z[i], color='red')
elif iris_y[i] == 1:
ax.scatter(x[i], y[i], z[i], color='orange')
else:
ax.scatter(x[i], y[i], z[i], color='gray')
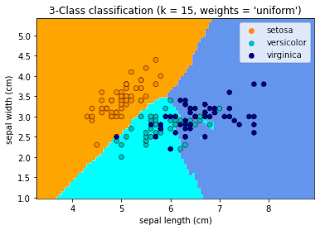
k最近傍法
最も単純なアルゴリズムがk最近傍法です。未知のデータを与えた場合、学習に使ったデータと比較して特徴が近いデータと同じ予測値を出力します。
詳細はこちら。今後ブログにもしていく予定です。
トレーニングセットとテストセット
AIモデルを作成する際は、未知のデータでの性能を考慮しません。しかし、学習データを使ってAIモデルを作成するという性質上、学習データを使って予測した結果を評価すると不当に高い性能となってしまいます。正当な評価をするためには未知のデータが必要です。しかし、未知のデータとセットで正しい予測結果のデータも必要となると、タスクによってはデータの収集が難しいことがあります。そのようなことから、既存のデータをトレーニングデータとテストデータで分割することが多くなっています。
KNN (k 最近傍法) での分類の例
※出し方はこちら# アヤメのデータをトレーニングデータとテストデータに分割
# 分割する前に配列をランダムに並べ替えています。
np.random.seed(0)
indices = np.random.permutation(len(iris_X))
#後ろの10個のデータをテストデータとして使用
iris_X_train = iris_X[indices[:-10]]
iris_y_train = iris_y[indices[:-10]]
iris_X_test = iris_X[indices[-10:]]
iris_y_test = iris_y[indices[-10:]]
# Create and fit a nearest-neighbor classifier
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier()
knn.fit(iris_X_train, iris_y_train)
#KNeighborsClassifier()
knn.predict(iris_X_test)
#array([1, 2, 1, 0, 0, 0, 2, 1, 2, 0])
iris_y_test
#array([1, 1, 1, 0, 0, 0, 2, 1, 2, 0])
次元の呪い
k近傍法に限らず、特長量が増えると計算量がグッと増えます。例えばk近傍法であれば、近くのデータとの距離を計算しています。2次元の特長量であれば大したことありませんが、これが10次元100次元となると計算量が指数関数的に増加してしまいます。そのような問題のことを次元の呪いと呼びます。
線形モデル: 回帰からスパースへ
糖尿病データセット
糖尿病データセットは、442 人の患者で測定された 10 の生理学的変数 (年齢、性別、体重、血圧) と、1 年後の疾患進行の指標で構成されています。
#後ろの20個のデータをテストデータとして分割
diabetes_X, diabetes_y = datasets.load_diabetes(return_X_y=True)
diabetes_X_train = diabetes_X[:-20]
diabetes_X_test = diabetes_X[-20:]
diabetes_y_train = diabetes_y[:-20]
diabetes_y_test = diabetes_y[-20:]
本課題は生理学的データから糖尿病の進行状況を予測するものです。
線形回帰
LinearRegressionは、目的変数として二乗誤差を最小とすることを目標に線形モデルのパラメータを調整します。
以下のグラフの描き方はこちら。
from sklearn import linear_model
regr = linear_model.LinearRegression()
regr.fit(diabetes_X_train, diabetes_y_train)
#LinearRegression()
print(regr.coef_)
#[ 0.30349955 -237.63931533 510.53060544 327.73698041 -814.13170937
# 492.81458798 102.84845219 184.60648906 743.51961675 76.09517222]
# 平均二乗誤差
np.mean((regr.predict(diabetes_X_test) - diabetes_y_test)**2)
#2004.5...
#スコアを求めます。0~1の間の数値で、1は完璧に予測できていることを表し、0は全く予測できていなことを表します。
regr.score(diabetes_X_test, diabetes_y_test)
#0.585...
正則化(縮小推定)
特長量の次元の数に対して、データの数が少ないと学習データに予測結果がフィットしすぎてしまう、過学習という現象が起こります。
例えば、以下のコードのように1つの特長量に対して、2つのデータだけで推論することを考えます。データというのは誤差が入り込みます。本来の予測として
の回帰式が正しいものとしたときに、適当な誤差を追加したデータで回帰式を作成するとグレーのようなグラフになります。
また、小さい誤差によって回帰式の変化も大きくなってしまいます。
※グラフの画像の作成はこちら
X = np.c_[ .5, 1].T
y = [.5, 1]
test = np.c_[ 0, 2].T
regr = linear_model.LinearRegression()
import matplotlib.pyplot as plt
plt.figure()
#<...>
np.random.seed(0)
for _ in range(6):
this_X = .1 * np.random.normal(size=(2, 1)) + X
regr.fit(this_X, y)
plt.plot(test, regr.predict(test))
plt.scatter(this_X, y, s=3)
#LinearRegression...
このような過学習に対する解決策として、回帰係数を小さくするという手法があります。その手法のことをリッジ回帰(Ridge)と呼びます。
regr = linear_model.Ridge(alpha=.1)
plt.figure()
#<...>
np.random.seed(0)
for _ in range(6):
this_X = .1 * np.random.normal(size=(2, 1)) + X
regr.fit(this_X, y)
plt.plot(test, regr.predict(test))
plt.scatter(this_X, y, s=3)
#Ridge...
alphaを、大きくするほどバイアスは高くなり、予測結果のばらつきは減ります。
例えば、以下のように糖尿病データセットを使用することで、汎化性能を調整することができます。
#array([0.0001, 0.00039811, 0.00158489,
0.00630957, 0.02511886,0.1])
alphas = np.logspace(-4, -1, 6)
print([regr.set_params(alpha=alpha)
.fit(diabetes_X_train, diabetes_y_train)
.score(diabetes_X_test, diabetes_y_test)
for alpha in alphas])
#[0.4725523580192521, 0.47248350131491657, 0.47220902528656805, 0.47111084422404537, 0.46665913731369846, 0.4480436728106735]
ノート:AIモデルが未知のデータを予測した時の性能が、学習データを予測した時と比べ著しく下がってしまうことを過学習と呼びます。また、リッジ回帰によって導入されたバイアスは、正則化と呼ばれます。
今回は直線の回帰式だったためわかりにくいですが、これが多項式回帰で波をうつような式だった場合リッジ回帰によって波が小さくなります。
スパース性
※グラフの作成はこちら。
特長量1つ目と2つ目のみでAIモデルを作成し可視化
ノート:糖尿病データセットの学習データは10の特長量を持ちます。10次元のデータは可視化できないので直感的に理解するのは難しいです。※原文の空白のスペースという訳の意味が不明でした。。。
以下のコードで1つ目と2つ目の特長量に対しての回帰係数を表示できます。
ols.coef_
#array([305.05479721, 10.44934916])
回帰係数を比較してみると、1つ目の方が大きく予測したい値に関わっていることがわかります。(少し増加するだけで、値の変化が大きくなるため)
特長量が多くて起こる次元の呪いを避けるために、有効な特長量のみ選択し、2つ目の特長量のようなあまり結果に関係しない特長量を切り捨てる手法があります。リッジ回帰と呼ばれる手法では、特長量を切り捨てることはせず全体の特長量の回帰係数を小さくします。ラッソ回帰と呼ばれる手法では、回帰係数を0にする特長量をいくつか用意し次元の呪いを回避します。このような手法を特にスパースと呼んだりします。
以下のコードでは実際にラッソ回帰で、糖尿病患者データのAIモデルを作成しています。結果をみると、1つ目の特長量と6つ目の特長量の回帰係数が0になっており、結果には影響が少ないことがわかります。
regr = linear_model.Lasso()
scores = [regr.set_params(alpha=alpha)
.fit(diabetes_X_train, diabetes_y_train)
.score(diabetes_X_test, diabetes_y_test)
for alpha in alphas]
best_alpha = alphas[scores.index(max(scores))]
regr.alpha = best_alpha
regr.fit(diabetes_X_train, diabetes_y_train)
#Lasso(alpha=0.025118864315095794)
print(regr.coef_)
#[ 0. -212.4... 517.2... 313.7... -160.8...
# -0. -187.1... 69.3... 508.6... 71.8... ]
同じ問題に対する複数のアプローチ方法
ある問題に対して、どのアルゴリズムがフィットするかはわかりません。
データセットが大規模な場合は、リッジ回帰も使用することができます。scikit-learnでは大規模なデータセットでもラッソ回帰が使用できるようなオブジェクトも提供しています。ラッソ回帰は特に特長量の数に対してデータの数が少ない問題に対して有効です。
分類
アヤメの分類のような、分類の問題で線形回帰をそのまま使用すると、予測した値が求めたい値とは全く別の値が出てしまいます。例えば、0か1のクラスで予測したいのに、0.1などが出力されてしまいます。そのため、分類にそのまま線形回帰のタスクを使うのは適切なアプローチではありません。
線形回帰の考えを分類の問題に適応させるためには、ロジスティック回帰かシグモイド関数を使用します。
以下のグラフを出力するコードはこちら。
黒い点がロジスティック回帰を使った予測結果。
青い線が線形回帰で作成した直線。
赤い線は青い線の結果をシグモイド関数に入力し分類問題に適応させた結果。
ロジスティック回帰を使用して分類を行うとこのような結果が得られます。
以下のグラフを出力するコードはこちら。
多クラス分類
予測するクラスが複数ある場合、よく使用される方法は1対全てのクラスのAIモデルを作成し最終的に多数決でクラスを決定する方法です。
正則化
ロジスティック回帰のハイパーパラメータで正則化の強度と種類を調整できます。Cが正則化項の強度で数値で指定します。値が小さいほど正則化項が強くなります。penaltyで種類を決定します。
none: ペナルティは追加されません。
l2: L2 ペナルティ項を追加します。これがデフォルトの選択です。
l1: L1 ペナルティ項を追加します。
elasticnet: L1 と L2 の両方のペナルティ項が追加されます。
練習
最近傍法とロジスティック回帰を使用して、手書き文字のデータセットを分類してみましょう。最後の10%をテストデータとしてスコアを確認してみましょう。
from sklearn import datasets, neighbors, linear_model
X_digits, y_digits = datasets.load_digits(return_X_y=True)
X_digits = X_digits / X_digits.max()
n_samples = len(X_digits)
X_train = X_digits[: int(0.9 * n_samples)]
y_train = y_digits[: int(0.9 * n_samples)]
X_test = X_digits[int(0.9 * n_samples) :]
y_test = y_digits[int(0.9 * n_samples) :]
knn = neighbors.KNeighborsClassifier()
logistic = linear_model.LogisticRegression(max_iter=1000)
print("KNN score: %f" % knn.fit(X_train, y_train).score(X_test, y_test))
print(
"LogisticRegression score: %f"
% logistic.fit(X_train, y_train).score(X_test, y_test)
)
#KNN score: 0.961111
#LogisticRegression score: 0.933333
サポートベクターマシン (SVM)
線形SVM
サポートベクターマシンは分類用のアルゴリズムです。サポートベクターマシンは、二つのクラスの間隔を最大化するように境界線を引いて分類を行うアルゴリズムです。下記の画像であれば、黒の実線を境界に、赤と青のクラスに分類をしています。赤と青の点で強調されている点がありますが、それらからの距離が最大になるように境界線を引きます。
正則化はパラメータCを用いて設定されます。Cは0以上の値で、値が小さいほど正則化の強さが大きくなります。
グラフを書くためのコードはこちら。
正則化されていないグラフ(C=1)
正則化を加えたグラフ(C=0.05)
※参考にしたページには下のグラフがデフォルトと表記されていますが、SVMでハイパーパラメータを設定しないとC=1となるはずなのでデフォルトは上のグラフになると思います。謝っていたら指摘してください。
例:アヤメのデータセットをSMVを使って分類する
データはこちら。
SVMは分類であればSVC、考え方を応用し回帰に使用できるようにしたSVRで使用することができます。
今回は分類のタスクですので、SVCを使用します。
from sklearn import svm
from sklearn.datasets import load_iris
'''
#データをロード済みの場合はコメントアウトしたまま。
iris_X, iris_y = datasets.load_iris(return_X_y=True)
# 分割する前に配列をランダムに並べ替えています。
np.random.seed(0)
indices = np.random.permutation(len(iris_X))
#後ろの10個のデータをテストデータとして使用
iris_X_train = iris_X[indices[:-10]]
iris_y_train = iris_y[indices[:-10]]
iris_X_test = iris_X[indices[-10:]]
iris_y_test = iris_y[indices[-10:]]
'''
svc = svm.SVC(kernel='linear')
svc.fit(iris_X_train, iris_y_train)
#SVC(kernel='linear')
注意:データの正規化 SVMを含む多くのアルゴリズムは、各特徴量を正規化、標準化しなければ適切な予測を行うことができません。
カーネルの使用
クラスは必ず直線、平面で分離できるとは限りません。SVMでは、そのような時でも分離が可能になるように既存の特徴量を計算し、新たな特徴量を増やします。特徴量を増やすことにより、例えば2次元を3次元にすることにより線形分離可能になることがあります。これを線形分離可能になるまで繰り返します。詳細は本ブログのこちらの記事でも解説しています。
どのように特徴量を増やすのかによって、境界の引き方も代わります。その特徴量の増やし方のことをカーネル関数と呼びます。カーネル関数もSVMのハイパーパラメータとして指定します。
実際にカーネルを関数を変えた時のグラフが次になります。
※グラフを書くコードはこちら。
線形カーネル関数
svc = svm.SVC(kernel='linear')
多項式カーネル関数
svc = svm.SVC(kernel='poly',degree=3)
# degree: 多項式の次数
RBFカーネル関数
svc = svm.SVC(kernel='rbf')
# gamma: 簡単に言うと境界の複雑さを表すハイパーパラメータです。
# 大きいほど境界線が複雑になります。⇨過学習を起こしやすくなる。
こちらを使用すると、GUI操作でハイパーパラメータの操作を行うことができます。
練習
アヤメのデータセットの最初の2つの特徴を使用して、クラス1と2を分類してみましょう。各クラスの10%を除外しテストデータとし、テストデータでスコアを求めてみましょう。
注意:元のデータの下位10% をテストデータとすると1つのデータでしかテストできません。(クラス順にデータが並んでいるため)トレーニングデータを分割するときは注意しましょう。
参考解答例:元はこちら
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets, svm
iris = datasets.load_iris()
X = iris.data
y = iris.target
X = X[y != 0, :2]
y = y[y != 0]
n_sample = len(X)
np.random.seed(0)
order = np.random.permutation(n_sample)
X = X[order]
y = y[order].astype(float)
X_train = X[: int(0.9 * n_sample)]
y_train = y[: int(0.9 * n_sample)]
X_test = X[int(0.9 * n_sample) :]
y_test = y[int(0.9 * n_sample) :]
# fit the model
for kernel in ("linear", "rbf", "poly"):
clf = svm.SVC(kernel=kernel, gamma=10)
clf.fit(X_train, y_train)
plt.figure()
plt.clf()
plt.scatter(
X[:, 0], X[:, 1], c=y, zorder=10, cmap=plt.cm.Paired, edgecolor="k", s=20
)
# Circle out the test data
plt.scatter(
X_test[:, 0], X_test[:, 1], s=80, facecolors="none", zorder=10, edgecolor="k"
)
plt.axis("tight")
x_min = X[:, 0].min()
x_max = X[:, 0].max()
y_min = X[:, 1].min()
y_max = X[:, 1].max()
XX, YY = np.mgrid[x_min:x_max:200j, y_min:y_max:200j]
Z = clf.decision_function(np.c_[XX.ravel(), YY.ravel()])
# Put the result into a color plot
Z = Z.reshape(XX.shape)
plt.pcolormesh(XX, YY, Z > 0, cmap=plt.cm.Paired)
plt.contour(
XX,
YY,
Z,
colors=["k", "k", "k"],
linestyles=["--", "-", "--"],
levels=[-0.5, 0, 0.5],
)
#スコアを表示
print(kernel,":",clf.score(X_train, y_train))
plt.title(kernel)
plt.show()